連鎖ランダムフォレストで欠損値を代入する「missRanger」パッケージの紹介です。欠損値の作成、代入に便利なパッケージです。
パッケージバージョンは2.2.0。実行コマンドはR version 4.2.2で確認しています。
パッケージのインストール
下記、コマンドを実行してください。
#パッケージのインストール
install.packages("missRanger")実行コマンド
詳細はコメント、パッケージのヘルプを確認してください。
#パッケージの読み込み
library("missRanger")
###データ例の作成#####
set.seed(1234)
n <- 10
TestData <- data.frame(Group = sample(paste0("Group ", 1:2), n, replace = TRUE),
Time_1 = round(rnorm(n) - 1.5, 2),
Time_2 = round(rnorm(n), 2),
Time_3 = round(rnorm(n) - 1.5, 2))
TestData[1:4, 2:4] <- sample(1:2, 12, replace = TRUE)
########
#データに欠損値を代入する:generateNAコマンド
#データを指定:xオプション;vector,matrix,data.frameが指定可能
#列ごとの欠損値の代入確率:pオプション;0.1-1.0の範囲
#seedの固定:seedオプション
ResultData <- generateNA(x = TestData, p = 0.3, seed = 1234)
#実行結果
ResultData
# Group Time_1 Time_2 Time_3
#1 Group 2 2.00 2.00 2.00
#2 Group 2 1.00 NA 1.00
#3 Group 2 1.00 2.00 1.00
#4 Group 2 1.00 NA 2.00
#5 <NA> NA 2.42 -2.44
#6 <NA> NA 0.13 NA
#7 Group 1 -2.50 NA NA
#8 Group 1 -2.28 -0.44 -2.21
#9 Group 1 NA 0.46 -2.00
#10 <NA> -0.54 -0.69 NA
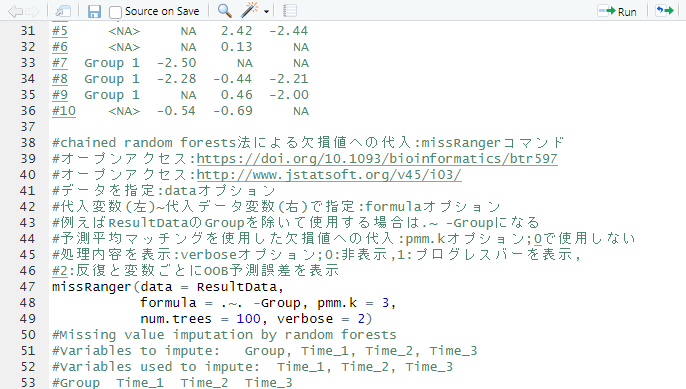
#chained random forests法による欠損値への代入:missRangerコマンド
#オープンアクセス:https://doi.org/10.1093/bioinformatics/btr597
#オープンアクセス:http://www.jstatsoft.org/v45/i03/
#データを指定:dataオプション
#代入変数(左)~代入データ変数(右)で指定:formulaオプション
#例えばResultDataのGroupを除いて使用する場合は.~ -Groupになる
#予測平均マッチングを使用した欠損値への代入:pmm.kオプション;0で使用しない
#処理内容を表示:verboseオプション;0:非表示,1:プログレスバーを表示,
#2:反復と変数ごとにOOB予測誤差を表示
missRanger(data = ResultData,
formula = .~. -Group, pmm.k = 3,
num.trees = 100, verbose = 2)
#Missing value imputation by random forests
#Variables to impute: Group, Time_1, Time_2, Time_3
#Variables used to impute: Time_1, Time_2, Time_3
#Group Time_1 Time_2 Time_3
#iter 1: 1.0000 1.0000 0.9862 1.6359
#
# Group Time_1 Time_2 Time_3
#1 Group 2 2.00 2.00 2.00
#2 Group 2 1.00 2.42 1.00
#3 Group 2 1.00 2.00 1.00
#4 Group 2 1.00 -0.69 2.00
#5 Group 2 2.00 2.42 -2.44
#6 Group 2 2.00 0.13 1.00
#7 Group 1 -2.50 -0.44 2.00
#8 Group 1 -2.28 -0.44 -2.21
#9 Group 1 1.00 0.46 -2.00
#10 Group 2 -0.54 -0.69 -2.21少しでも、あなたの解析が楽になりますように!!