「データを解析する前におこなっている作業の紹介」を先日紹介しました。そんな、解析する前にデータの特徴を探るのに便利なコマンドが収録されたパッケージの紹介です。
データを解析する前の作業を紹介
https://www.karada-good.net/analyticsr/r-293/
多くのコマンドの中から、これは便利と感じた「素数、最大公約数、フィボナッチ数、要素の組み合わせ」と「データの特徴を把握しながら図式化する」コマンドを紹介します。その他のコマンドはパッケージヘルプを確認していただければと思います。
パッケージのバージョンは0.99.44。 windows11のR version 4.1.2で確認しています。
パッケージのインストール
下記、コマンドを実行してください。
#パッケージのインストール
install.packages("DescTools")実行コマンド
詳細はコメント、パッケージのヘルプを確認してください。
#パッケージの読み込み
library("DescTools")
#nまでの素数の表示:Primeコマンド
Primes(n = 37)
[1] 2 3 5 7 11 13 17 19 23 29 31 37
#素数であるかの確認:IsPrimeコマンド
IsPrime(Primes(n = 37))
[1] TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE
#最少公倍数の計算:LCMコマンド
LCM(2, 3)
[1] 6
#最大公約数の計算:GCDコマンド
GCD(160, 25)
[1] 5
#フィボナッチ数:Fibonacciコマンド
Fibonacci(1:6)
[1] 1 1 2 3 5 8
#要素の全組み合わせ:Permnコマンド
Permn(c(1:3))
[,1] [,2] [,3]
[1,] 1 2 3
[2,] 2 1 3
[3,] 2 3 1
[4,] 1 3 2
[5,] 3 1 2
[6,] 3 2 1
#組み合わせの数を計算:factorialコマンド
factorial(length(c(1:3)))
[1] 6
#要素範囲を指定した全組み合わせ:CombSetコマンド
CombSet(c(1:3), 2, repl = FALSE, ord = TRUE)
[,1] [,2]
[1,] 1 2
[2,] 2 1
[3,] 1 3
[4,] 3 1
[5,] 2 3
[6,] 3 2
#2つのベクトルの組み合わせ:CombPairsコマンド
CombPairs(c(1:3), c(3:1))
Var1 Var2
1 1 3
2 2 3
3 3 3
4 1 2
5 2 2
6 3 2
7 1 1
8 2 1
9 3 1
###データ例の作成#####
n <- 1000
TestData <- data.frame(Group = sample(c(paste0("Group", 1:3), NA), n, replace = TRUE),
Data1 = sample(1:200, n, replace = TRUE),
Data2 = rnorm(n),
Data3 = factor(sample(c("YES", "NO", NA), n, replace = TRUE)))
########
#因子の分布を確認:Descコマンド
#カテゴリ
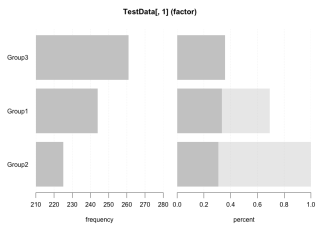
Desc(TestData[, 1], plotit = TRUE, digits = 3)
TestData[, 1] (factor)
length n NAs levels unique dupes
1'000 780 220 3 3 y
level freq perc cumfreq cumperc
1 Group3 3e+02 3.6e+01% 3e+02 3.6e+01%
2 Group1 2e+02 3.2e+01% 5e+02 6.8e+01%
3 Group2 2e+02 3.2e+01% 8e+02 1.0e+02%
#連続変数
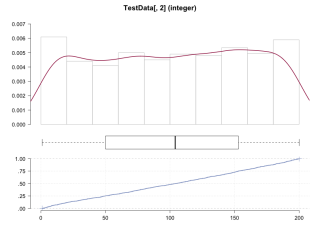
Desc(TestData[, 2], plotit = TRUE)
TestData[, 2] (integer)
length n NAs unique 0s mean meanSE
1e+03 1e+03 0 2e+02 0 1.03e+02 1.80e+00
.05 .10 .25 median .75 .90 .95
1.30e+01 2.40e+01 5.30e+01 1.04e+02 1.51e+02 1.79e+02 1.90e+02
range sd vcoef mad IQR skew kurt
1.99e+02 5.69e+01 5.55e-01 7.26e+01 9.80e+01 -4.49e-02 -1.20e+00
lowest : 1e+00 (4e+00), 2e+00 (5e+00), 3e+00 (7e+00), 4e+00 (2e+00), 5e+00 (2e+00)
highest: 2e+02 (7e+00), 2e+02 (3e+00), 2e+02 (8e+00), 2e+02 (6e+00), 2e+02 (4e+00)
#因子
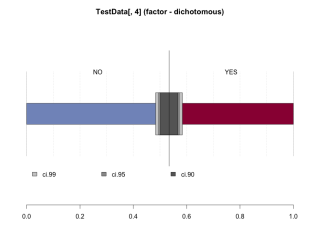
Desc(TestData[, 4], plotit = TRUE)
TestData[, 4] (factor - dichotomous)
length n NAs unique
1'000 661 339 2
freq perc lci9.50e-01 uci9.50e-01'
NO 3e+02 5.3e+01% 4.9e+01% 5.7e+01%
YES 3e+02 4.7e+01% 4.3e+01% 5.1e+01%
' 95%-CI Wilson
#データを箱ひげ図で確認
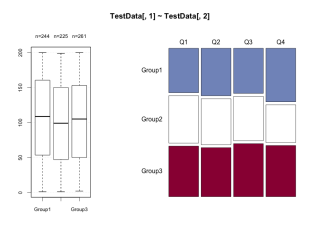
Desc(TestData[, 1] ~ TestData[, 2], plotit = TRUE)
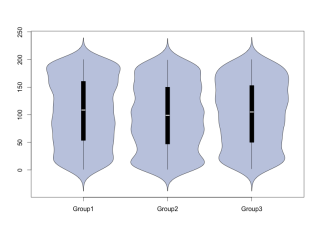
#バイオリンプロット:PlotViolinコマンド
PlotViolin(TestData[, 2] ~ TestData[, 1],
data = TestData, col = SetAlpha(hblue,0.5))
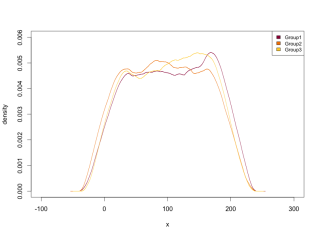
#密度グラフの作成:PlotMultiDensコマンド
PlotMultiDens(TestData[, 2] ~ TestData[, 1],
data = TestData)
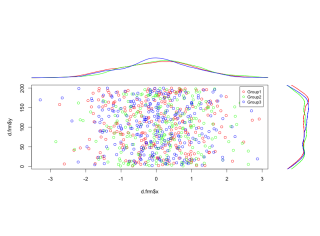
#散布図と密度グラフの同時描写:PlotMarDensコマンド
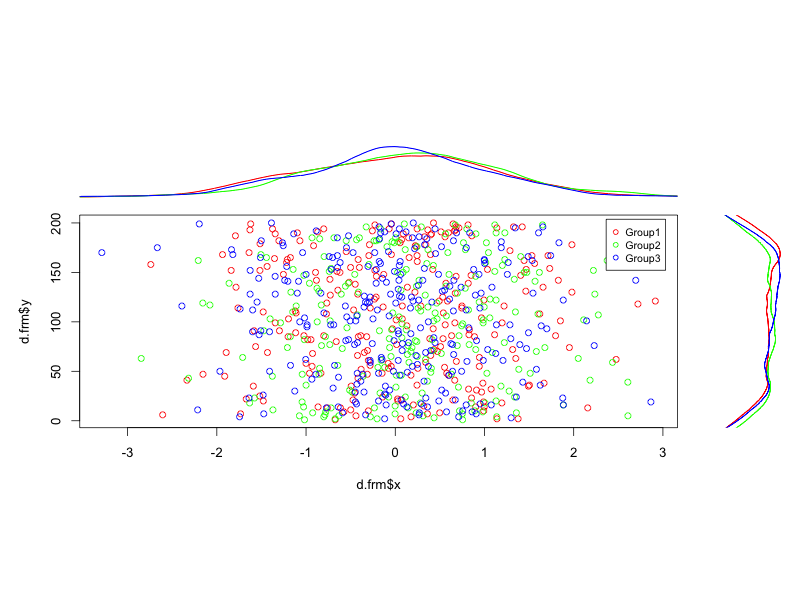
#グループを指定:grpオプション
PlotMarDens(y = TestData[, 2], x = TestData[, 3], grp = TestData[, 1])出力例
Descコマンドによる出力です。
・カテゴリ
・連続変数
・因子
・データを箱ひげ図で確認
・PlotViolinコマンド
・PlotMultiDensコマンド
・PlotMarDensコマンド
少しでも、あなたの解析が楽になりますように!!