factor classで不要な水準(Levels)が残っていると、図の描写などで余計なラベルが表示され邪魔な場合があります。そんな時には「droplevels」コマンドを使用してみてはいかがでしょうか。
実行コマンドはR version 4.2.2で確認しています。
実行コマンド
詳細はコメント、コマンドのヘルプを確認してください。
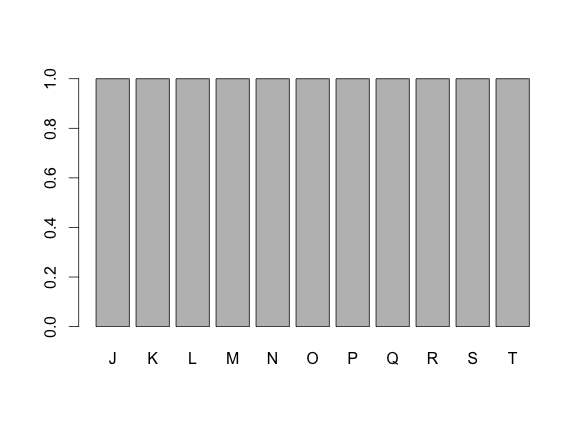
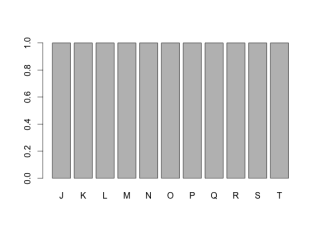
#因子の作成 TestFactor <- factor(1:24, labels = LETTERS[1:24]) TestFactor [1] A B C D E F G H I J K L M N O P Q R S T U V W X Levels: A B C D E F G H I J K L M N O P Q R S T U V W X #例えば10:20の位置を抽出 GetFactor <- TestFactor[10:20] GetFactor [1] J K L M N O P Q R S T Levels: A B C D E F G H I J K L M N O P Q R S T U V W X #水準(Levels)はそのまま,何かと不便な場合があります #例えばプロット,余計なx軸ラベルが残ります #そのままプロット plot(GetFactor) #不要な水準(Levels)を取り除く:droplevelsコマンド plot(droplevels(GetFactor))
出力結果
・そのままプロット
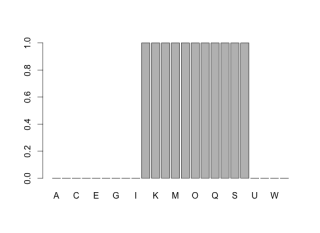
・droplevelsコマンド
少しでも、あなたのウェブや実験の解析が楽になりますように!!