ハッセ図は順序関係や群を理解するのに有効な方法だと思います。ハッセ図を描写する「hasseDiagram」パッケージを紹介します。ハッセ図は非常にシンプルな思想だと思います。
秋の夜長に美しいハッセ図を見るために、generateRandomDataコマンドを実行するのも風流かもしれません。もしかすると、ガロアのような発想が生まれるかも。
パッケージのバージョンは0.2.0。R version 4.2.2でコマンドを確認しています。
パッケージのインストール
下記コマンドを実行してください。
#パッケージのインストール
install.packages("hasseDiagram")
#下記を事項してRgraphvizパッケージインストール後に
#Update all/some/none? [a/s/n]:と表示された場合はnを選択してください
source("https://bioconductor.org/biocLite.R")
biocLite("Rgraphviz")実行コマンド
詳細はコマンド、パッケージヘルプを確認してください。
#パッケージの読み込み
library("hasseDiagram")
#ハッセ図データ例の作成:generateRandomDataコマンド
#ノード数の指定:nrNodesオプション, グラフ数の指定:minGraphsオプション
#ハッセ図の複雑度:densityオプション;0で結合なし,1で一直線です
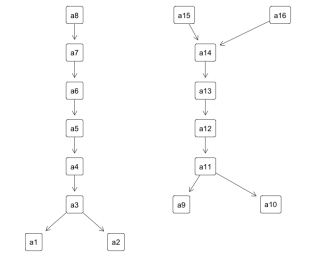
TestData <- generateRandomData(nrNodes = 16, minGraphs = 2, density = .7)
#データのプロット
hasse(TestData)
#オリジナルハッセ図のデータを作成
#マトリックスで指定します,[始点, 終点]の位置にTRUEで線(エッジ)を描写します
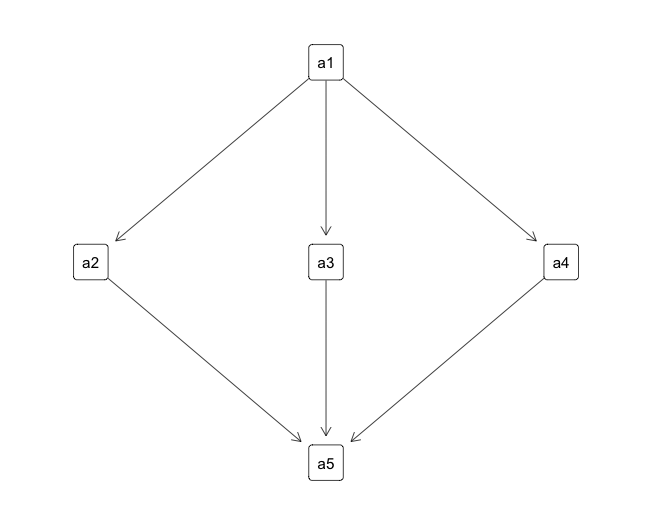
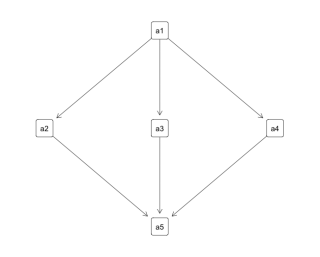
#ノード数5,"a1"から"a2, a3, a4"につなげ"a5"に集結します
#なお、グラフを分けてプロットする場合は分割したい列を全てFALSEにします
data <- matrix(data = FALSE, ncol = 5, nrow = 5)
#TRUEの代入
data[1, 2] <- data[1, 3] <- data[1, 4] <- TRUE
data[2, 5] <- data[3, 5] <- data[4, 5] <- TRUE
#データのプロット:hasseコマンド
hasse(data)
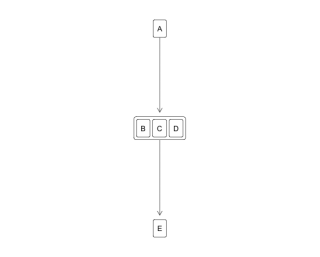
#同一レベルにあるノードをまとめてプロット
#[始点, 終点]が重複するようにTRUEを代入します
#TRUEの代入
data[2, 3] <- data[2, 4] <- data[3, 2] <-
data[3, 4] <- data[4, 2] <- data[4, 3] <- TRUE
#プロット:list(cluster = TRUE)オプション
#なお、ノードラベルはlabelsオプションで指定します
hasse(data, labels = c("A", "B", "C", "D", "E"), list(cluster = TRUE))出力例
・generateRandomDataコマンド
・オリジナルハッセ図のデータをプロット
・同一レベルにあるノードをまとめてプロット
少しでも、あなたの解析が楽になりますように!!