Rでは変数へのデータを代入する方法として「<-」を使用します。でもこの方法だと、むやみに変数が増える場合がありコードを改良する際に可読性に障害が出る場合があります。そこで、コードの可読性を向上するmagrittrパッケージをご紹介します。このパッケージを利用することで”<-“ではなく「%>%」(パイプ)でコードをつなげることで、可読性が向上するのでぜひ試してみてください。ただ、magrittrパッケージをインストール必要があるのが難点です。
「magrittr」パッケージの導入
下記コードを実行することで導入することができます。
参考URL: https://github.com/smbache/magrittr
#CRANからのインストール
install.packages("magrittr")
#githubからのインストール
library(devtools) #devtoolsが無い場合は右を実行:install.packages("devtools")
install_github(“smbache/magrittr")「%>%」(パイプ)の使用例
基本例とggplot2を組み込んだ例を示します。なお、基本例に示されているコードで出力される図は全て一緒です。
基本例です。従来の方法も参考までに示します。
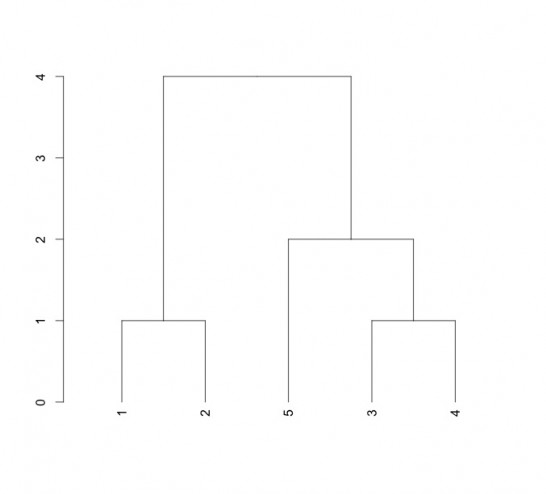
[code language="R"] #%>%を利用した例 c(1:5) %>% #データの準備 Dist(method = "euclidean") %>% #距離の計算 hclust(method = "complete") %>% #結合の設定 as.dendrogram %>% #デンドログラムクラスへの変換 plot #プロット #従来の方法 #例1 plot(as.dendrogram(hclust(Dist(c(1:5), method = "euclidean"), method = "complete"))) #例2 Data <- c(1:5) DistData <- Dist(Data, method = "euclidean") #距離の計算 HclustDistData <- hclust(DistData, method = "complete") #結合の設定 PlotData <- as.dendrogram(HclustDistData) #デンドログラムクラスへの変換 plot(PlotData) #プロット #こんな使い方も出来ます。 ShowPlot <- c(1:5) %>% #データの準備 Dist(method = "euclidean") %>% #距離の計算 hclust(method = "complete") %>% #結合の設定 as.dendrogram %>% #デンドログラムクラスへの変換 plot(ShowPlot) #プロット
ggplot2を組み込んだ例
library("magrittr")
library("ggplot2")
library("dplyr")
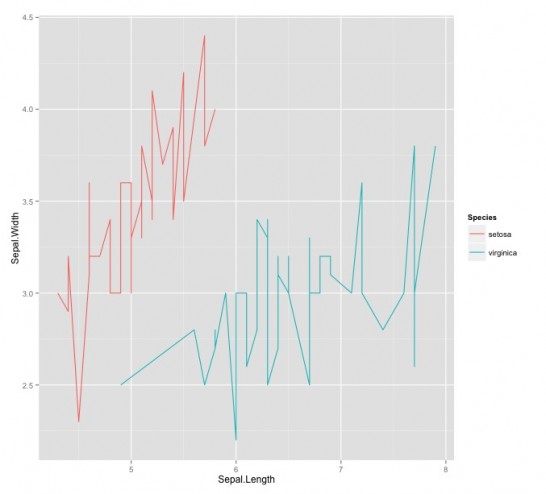
iris %>%
subset(iris[,5] == "setosa" | iris[,5] == "virginica") %>%
qplot(Sepal.Length, Sepal.Width, color = Species, data = ., geom = "line") %>%
print出力される図
基本例
ggplot2を組み込んだ例
少しでも、ウェブや実験の解析が楽になりますように!!
もし、不明点がありましたらこちらからお問い合わせください。