Circular Visualizationという表現方法をご存知でしょうか?遺伝子解析や社会学などで見かけるグラフで、項目を円に並べ線で結び関係性を把握する表現方法です。
そんなCircular Visualizationをお手軽に作成できる「circlizeパッケージ」をご紹介します。このパッケージはCIRCOSという団体がフリーで配布していて、natureやscienceだけでなく、New York Timesなどでも利用されています。
CIRCOS:http://circos.ca/
非常に有用なパッケージだと思います。ぜひ、ご利用ください。
「qicharts」パッケージの導入
下記コードを実行することで導入することができます。
URL: http://cran.r-project.org/web/packages/circlize/index.html
install.packages("circlize")コマンド例の紹介
行と列で項目が異なる場合と同じ場合の例をご紹介します。実行してデータを確認してみてください。
また、個のパッケージはいろいろできますので試してみてください。
まずは、パッケージの読み込みです。色を設定する為にRColorBrewerも読み込んでいます。
library("circlize")
library("RColorBrewer")行と列で項目が異なる場合
#色の準備
ColColor <- colorRampPalette(c("#2ca9e1", "#e7609e", "#f7c114", "#543f32")) #行の色
RowColor <- colorRampPalette(c("#ec6d51", "#3f312b", "#674196", "#82ae46")) #列の色
set.seed(123)
mat <- matrix(sample(1:100, 18, replace = TRUE), 3, 6)
rownames(mat) <- letters[1:3]
colnames(mat) <- LETTERS[1:6]
rn <- rownames(mat)
cn <- colnames(mat)
mat #データの確認
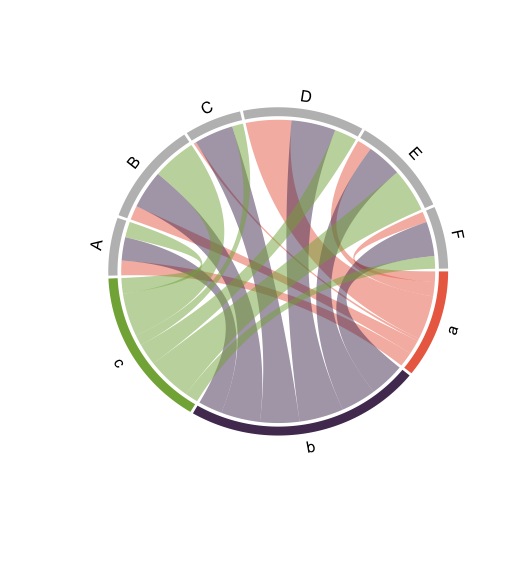
chordDiagram(mat,
transparency = 0.5, #透明度の調整
#column.col = ColColor(ncol(mat)), #行の色を設定、データ行で色数を指定
row.col = RowColor(nrow(mat)), #列の色を設定、データ列で色数を指定
grid.col = c(RowColor(nrow(mat)), rep("grey", ncol(mat))) #ラベルの色を設定、
#データ行で色を指定した場合はc(RowColor(ncol(mat)), rep("grey", nrow(mat)))
)行と列で項目が同じ場合
#色の準備
ColColor <- colorRampPalette(c("#2ca9e1", "#e7609e", "#f7c114", "#543f32")) #行の色
RowColor <- colorRampPalette(c("#ec6d51", "#3f312b", "#674196", "#82ae46")) #列の色
set.seed(123)
mat = matrix(sample(100, 25), 5)
rownames(mat) = letters[1:5]
colnames(mat) = letters[1:5]
#diag(mat) <- 0 #対角要素を0に置換、自己循環が邪魔な場合は使用してみてください。
mat #データの確認
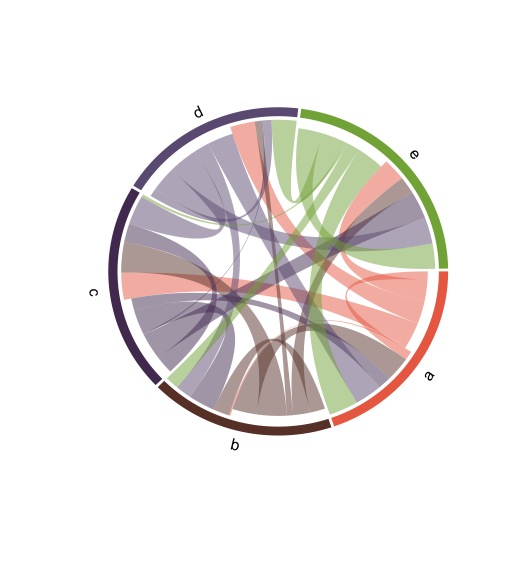
chordDiagram(mat,
directional = TRUE, #指向性を解りやすく表現
transparency = 0.5, #透明度の調整
row.col = RowColor(nrow(mat)), #列の色を設定、データ列で色数を指定
grid.col = RowColor(nrow(mat)) #ラベルの色を設定、
)出力される図
行と列で項目が異なる場合
行と列で項目が同じ場合
少しでも、あなたの解析が楽になりますように!!