NCBIデータベースをRから操作するパッケージはいくつか存在しますが、本パッケージはPubMedデータベースから情報を収集するのに特化したパッケージです。かなり簡単に情報を収集することが出来ます。
実行コマンドでは検索クエリに”r statistical software”を指定し、取得したデータをGoogleスプレッドシートへアップロードと「xlsx」ファイルで保存する例を紹介します。
パッケージバージョンは2.13。実行コマンドはR version 4.2.2で確認しています。
パッケージのインストール
下記、コマンドを実行してください。
#パッケージのインストール
install.packages("easyPubMed")実行コマンド
詳細はコメント、パッケージのヘルプを確認してください。
#パッケージの読み込み
library("easyPubMed")
#検索クエリを指定
#pubmedの検索タグが使用可能です
#参考:https://ja.wikipedia.org/wiki/MEDLINE
SetQuery <- "r statistical software"
#PubmedIdを取得:get_pubmed_idsコマンド
PubMedID <- get_pubmed_ids(pubmed_query_string = SetQuery)
#PubmedIdを元にデータを最大5,000まで取得:fetch_pubmed_dataコマンド
#取得開始の番号:retstartコマンド
#取得終了の番号:retmaxコマンド
#最新10の論文データを取得
#結果はXMLで取得
GetResult <- fetch_pubmed_data(pubmed_id_list = PubMedID,
retstart = 0, retmax = 10,
format = "xml", encoding = "UTF-8")
#fetch_pubmed_dataコマンドで取得した結果をdataframeにする:table_articles_byAuthコマンド
#著者の取得:included_authorsオプション;"first","last","all"
#取得するアブストラクトの文字数:max_charsオプション;-1で全て
#欠損値の処理:autofillオプション;TRUE/FALSE
#データを作業フォルダに出力:dest_fileオプション;NULLで出力無し
##出力する場合は「ファイル名.txt」を指定
#キーワードの出力:getKeywordsオプション;TRUE/FALSE
#エンコードの指定:encodingオプション
ResultFrame <- table_articles_byAuth(pubmed_data = GetResult,
included_authors = "last",
max_chars = -1,
dest_file = NULL,
encoding = "UTF-8")
###参考:Googleスプレッドシートへアップロード#####
#googlesheets4パッケージは下記URLの記事を参考にしてください
#https://www.karada-good.net/analyticsr/r-744/
#googlesheets4パッケージがなければインストール
if(!require("googlesheets4", quietly = TRUE)){
install.packages("googlesheets4");require("googlesheets4")
}
#Googleアカウントに接続:gs4_authコマンド
#既に接続済みであればアカウント選択のメッセージが表示されます
gs4_auth()
#データをアップロード
#新規Googleスプレッドシートを作成:gs4_createコマンド
gs4_create(name = "PubMedData",
sheets = list("PubMedData" = ResultFrame))
########
###参考:作業フォルダにxlsxで保存#####
#openxlsxパッケージは下記URLの記事を参考にしてください
#https://www.karada-good.net/analyticsr/r-535/
if(!require("openxlsx", quietly = TRUE)){
install.packages("openxlsx");require("openxlsx")
}
#Workbook objectの作成:createWorkbookコマンド
PubmedWb <- createWorkbook(creator = "PubMed",
title = "PubMed",
subject = "PubMed",
category = "PubMed")
#ワークシートの追加:addWorksheetコマンド
addWorksheet(wb = PubmedWb,
sheetName = "PubMed",
tabColour = "blue",
zoom = 80)
#シートにデータを書き込む:writeDataコマンド
writeData(wb = PubmedWb,
sheet = 1, x = ResultFrame,
startRow = 3, startCol = 2,
borderColour = "blue")
#abstractに含まれる特定キーワードで塗りつぶす
#セルのスタイル
CellStyle <- createStyle(bgFill = "red")
#範囲指定
conditionalFormatting(wb = PubmedWb, sheet = 1,
style = CellStyle,
cols = 5, rows = 3:nrow(ResultFrame),
type = "contains",
rule = "tumor-bearing mice")
#作業フォルダにxlsxファイルを保存:saveWorkbookコマンド
saveWorkbook(wb = PubmedWb,
file = "Pubmed.xlsx",
overwrite = TRUE)
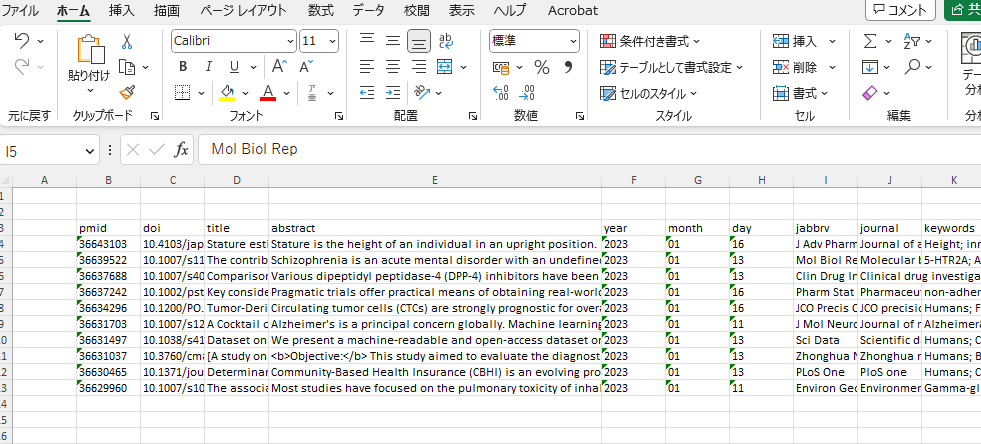
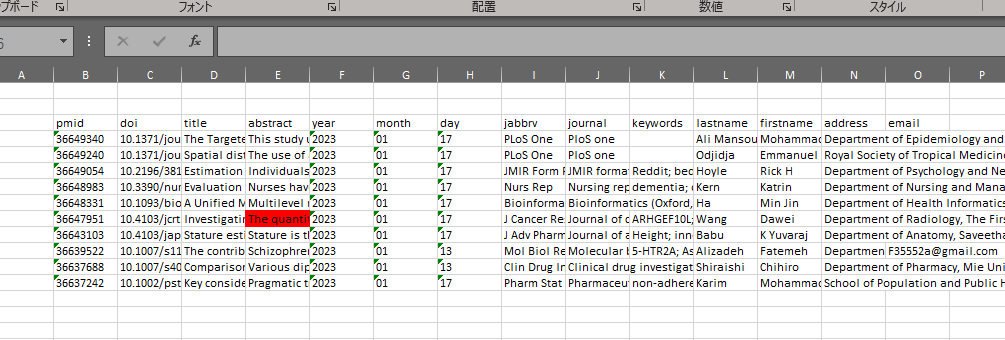
########出力例
・参考:Googleスプレッドシートへアップロード
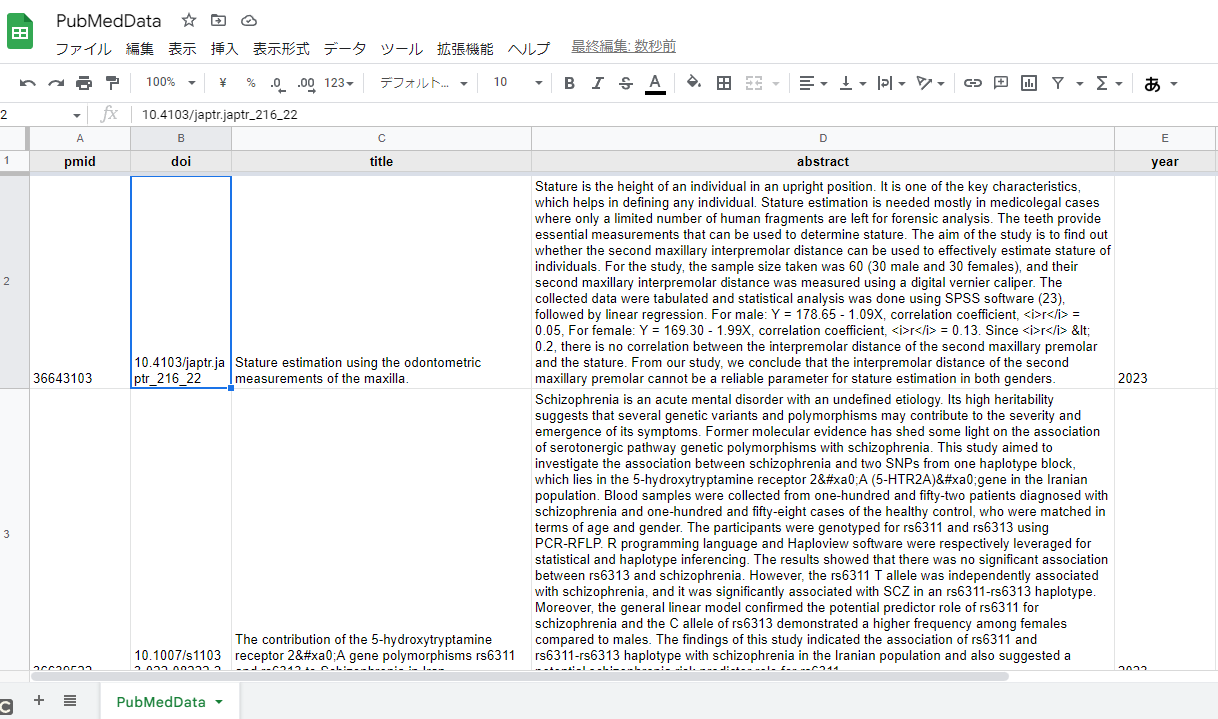
・参考:作業フォルダにxlsxで保存
少しでも、あなたの解析が楽になりますように!!