少ないコードでヒートマップを作成可能なパッケージの紹介です。細かい体裁などが得意で理想のヒートマップが作成できる「ComplexHeatmap」パッケージを利用するパッケージです。ですので、「ComplexHeatmap」パッケージのインストールが必要です。また、「ComplexHeatmap」パッケージと共通のコマンドを使用することができます。
「tidyHeatmap」パッケージを利用するとかなり、コード数を減らすことができるのでお勧めです。特にグループ分けがdplyr::group_byコマンドで可能なので大変便利です。
参考までに「ComplexHeatmap」パッケージの記事を紹介します。
パッケージのバージョンは1.8.1。実行コマンドはR version 4.2.2で確認しています。
パッケージのインストール
下記コマンドを実行してください。
#ComplexHeatmapパッケージがなければインストール
if (!require("BiocManager", quietly = TRUE)){
install.packages("BiocManager")
}
BiocManager::install("ComplexHeatmap", force = TRUE)
#パッケージのインストール
install.packages("tidyHeatmap")実行コマンド
詳細はコメント、パッケージヘルプを確認してください。
#パッケージの読み込み
library("tidyHeatmap")
###データ例の作成#####
#tidyverseパッケージがなければインストール
if(!require("tidyverse", quietly = TRUE)){
install.packages("tidyverse");require("tidyverse")
}
set.seed(1234)
n <- 300
TestData <- tibble(Group = factor(rep(paste0("Group_", 1:30),
each = 10),
level = paste0("Group_", 1:30)),
SubGroup = factor(rep(paste0("SubGroup_", 1:10),
time = 30),
level = paste0("SubGroup_", 1:30)),
Value = sample(c(1:100), n, replace = TRUE),
SubValue = sample(c(1:100), n, replace = TRUE),
Flag_1 = factor(rep(2:3, each = 10, time = 15)),
Flag_2 = factor(rep(c("OK", "NG"), each = 1,
time = 150)),
FlagValue_1 = rep(1:10, each = 10, time = 3),
FlagValue_2 = factor(rep(11:20, time = 30)),
FlagValue_3 = rep(1:10, each = 10, time = 3),
FlagValue_4 = rep(11:20, time = 30),
FlagValue_5 = rep(11:20, time = 30))
########
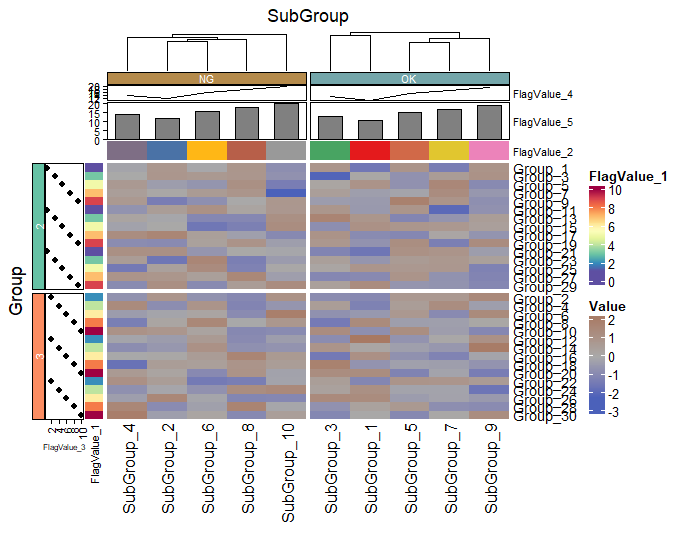
TestData %>%
#heatmapをグループで分割するときはgroup_byで設定
dplyr::group_by(Flag_1, Flag_2) %>%
#heatmapコマンド
heatmap(.row = Group, .column = SubGroup, .value = Value,
#データのスケール化;"none","row","column","both)
scale = "row",
#クラスタリングの適応有無
cluster_rows = FALSE, cluster_columns = TRUE,
#クラスタリング手法
clustering_distance_rows = "pearson",
#カラーパレットを指定
palette_value = c("#4b61ba", "#A9A9A9", "#a87963"),
#グループ分割の色を指定
palette_grouping = list(
#Flag_1の分割
c("#66C2A5", "#FC8D62"),
#Flag_2の分割
c("#b58b4c", "#74a6aa")
),
#セルの枠線:
#rect_gp = grid::gpar(col = "#161616", lwd = 0.5),
) %>%
#x,y方向にグラフを付与:add_point,add_bar,add_lineコマンド
#データ内容から付与方向は自動で判断
add_point(FlagValue_3, palette = NULL,
annotation_name_gp = grid::gpar(fontsize = 8)) %>%
add_line(FlagValue_4, palette = NULL,
size = unit(0.4, "cm")) %>%
add_bar(FlagValue_5, palette = NULL, size = unit(1, "cm"),
annotation_name_gp = grid::gpar(fontsize = 8)) %>%
#x,y方向にデータを付与:add_titleコマンド
#データ内容から付与方向は自動で判断
add_tile(FlagValue_1, show_legend = TRUE,
annotation_name_gp = grid::gpar(fontsize = 6)) %>%
add_tile(FlagValue_2, show_legend = FALSE)出力例
少しでも、あなたの解析が楽になりますように!!