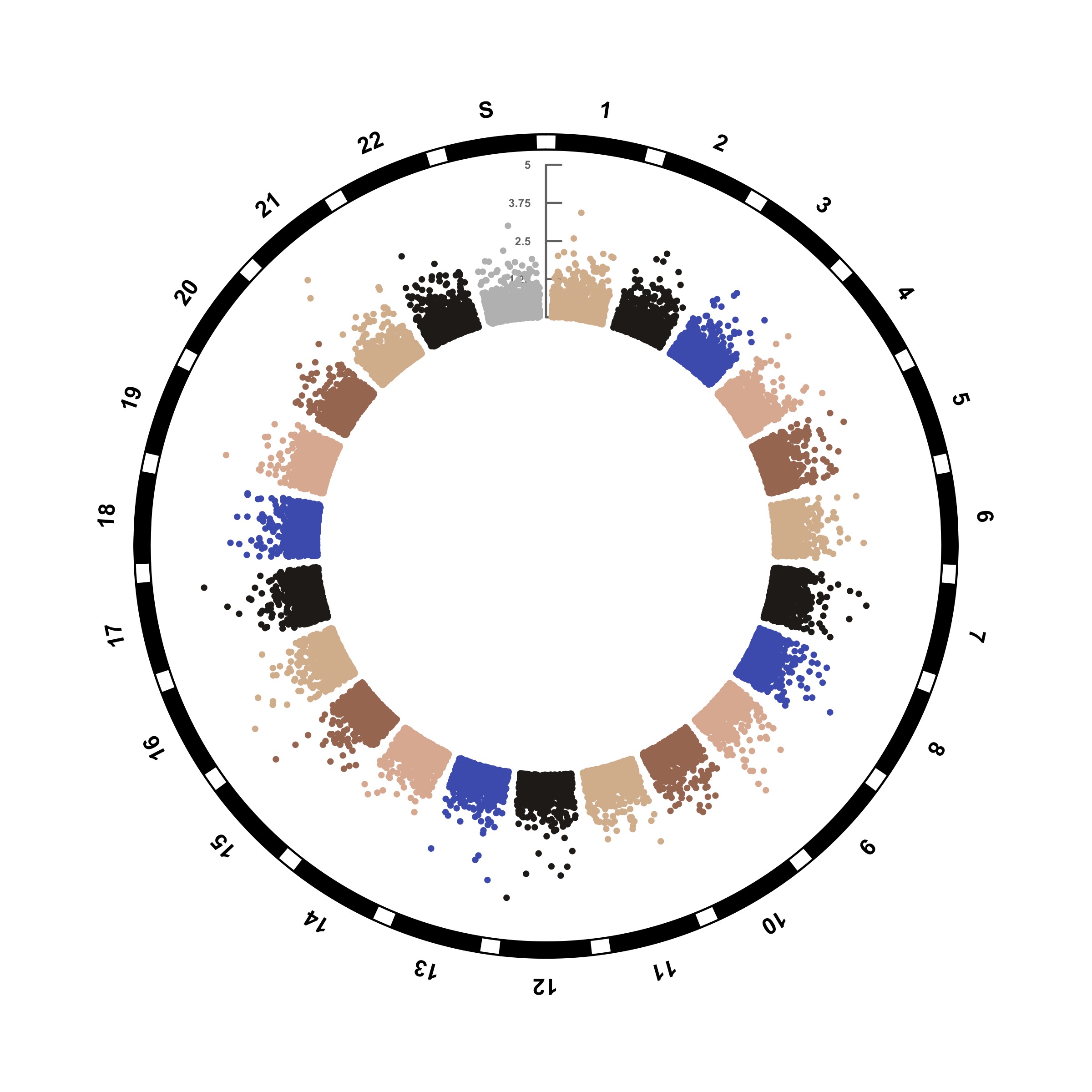
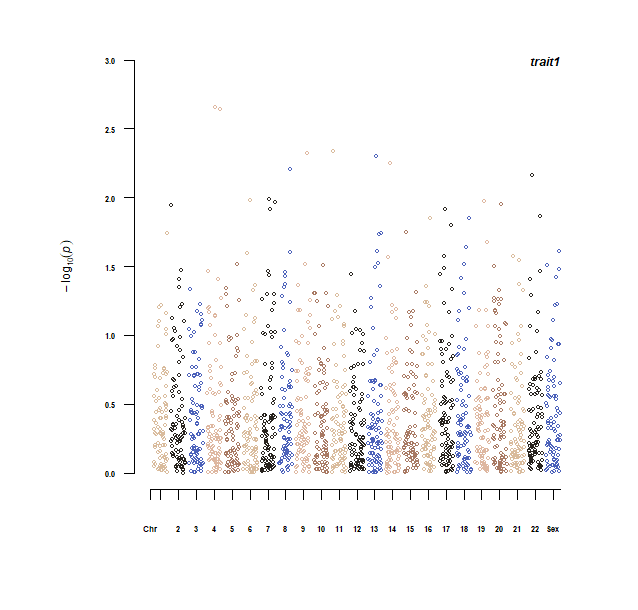
本パッケージはSNP結果を確認するのに便利なサークルプロット、マンハッタンプロット、QQplotをなどを作成できます。大変オプションが多いのでヘルプ参照を推奨します。
パッケージバージョンは4.0.0。実行コマンドはwindows 11のR version 4.1.3で確認しています。
パッケージのインストール
下記、コマンドを実行してください。
#パッケージのインストール
install.packages("CMplot")実行コマンド
本パッケージではプロット結果を作業ディレクトリに出力します。そのため、実行コマンドではtcltkパッケージを利用して保存フォルダを選択できるようにしています。詳細はコマンド、パッケージのヘルプを確認してください。
#パッケージの読み込み
library("CMplot")
library("tcltk")
#出力の保存先を指定
setwd(paste(as.character(tkchooseDirectory(title = "保存先を選択"), sep = "", collapse ="")))
###データ例の作成#####
set.seed(1234)
i <- 2000
TestData <- data.frame(SNP = rep(paste("TEST", seq(i), sep = "")),
Chromosome = rep(c(1:22, "Sex"), length = i),
Position = sample(1000:20000, i, replace = TRUE),
trait1 = runif(i))
########
#マンハッタンプロット,サークルプロット,QQplotを一度に作成:CMplotコマンド
#データを指定:Pmapオプション
#プロット内容を指定:plot.typeオプション;"m":マンハッタンプロット,
#"c":サークルプロット,"q":QQplot,"b":全プロット
#サークルプロットの外円とマンハッタンプロットの間隔を調整:cir.bandオプション;初期値1
#サークルプロットのマンハッタンプロットの高さを調整:Hオプション;初期値1
#サークルプロットの半径を調整:rオプション;初期値1
#マンハッタンプロットの表示領域を調整:multracksオプション;初期値TRUE
#プロットの色を指定:colオプション
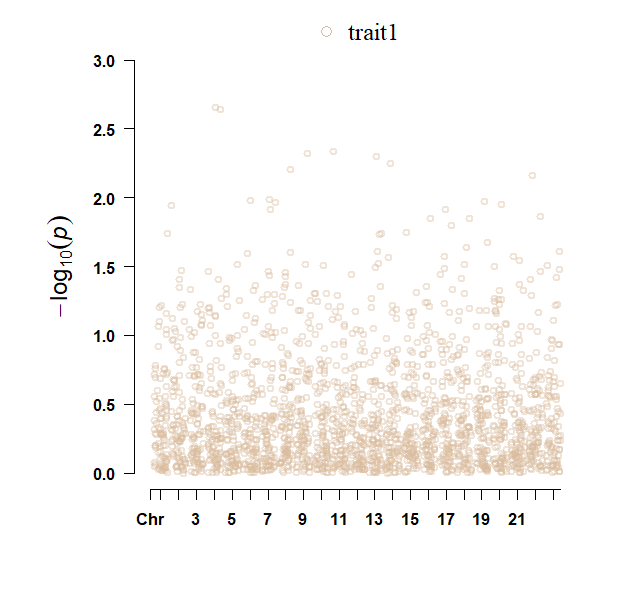
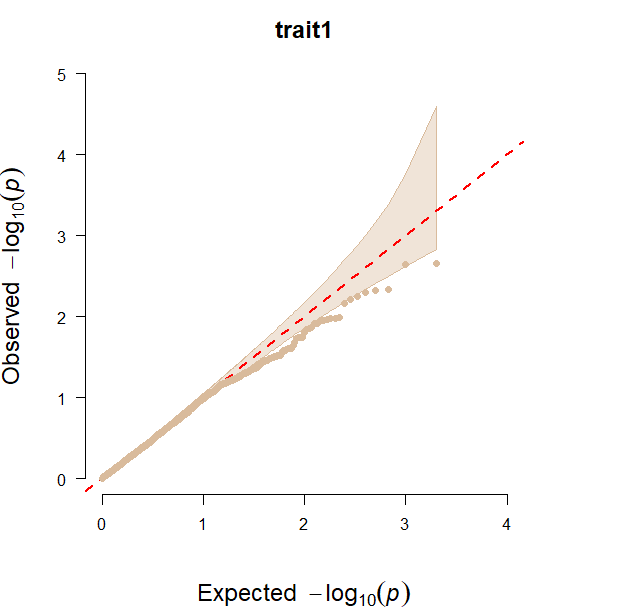
CMplot(Pmap = TestData, plot.type = "b", cir.band = 1, r = 3,
H = 2, multracks = TRUE, col = c("#d9bb9c", "#28231e", "#4b61ba", "#deb7a0", "#a87963"),
amplify = TRUE, file.output = FALSE, pch = 21)出力例
・SNP-Density Plotting

・Circular-Manhattan Plotting

・Multracks-Manhattan Plotting
・Multraits-Rectangular
・Multracks-QQ Plotting
少しでも、あなたの解析が楽になりますように!!